 |
|
| Appunti scientifiche |
|
 |
|
| Appunti scientifiche |
|
| Visite: 8307 | Gradito: |
Leggi anche appunti:Metodo delle velocità inizialiMetodo delle velocità iniziali Si determina la velocità all'inizio della Legge della conservazione della massa di Lavoisier (1789)Legge della conservazione della massa di Lavoisier (1789) 'In una reazione Acidi e basi secondo LewisAcidi e basi secondo Lewis La definizione più estensiva di acido e base |
 |
 |
L'importanza dell'analisi mendeliana dell'ereditarietà sta nel fatto che le leggi della segregazione e dell'assortimento indipendente dei fattori genetici (alleli) possono essere formulate anche senza sapere qual è la loro natura fisica e quali sono le strutture cellulari che li ospitano. I primi a rendersi conto che vi era uno stretto parallelismo tra il comportamento dei fattori genetici mendeliani ed i cromosomi furono nel 1902 lo statunitense Walter Sutton ed il tedesco Theodor Boveri. I cromosomi omologhi sono infatti due come i fattori genetici mendeliani, si separano durante la meiosi nei gameti (segregazione dei fattori genetici) ed i gameti si ricombinano casualmente durante la fecondazione. L'ipotesi che le due forme alleliche di un gene fossero portate dai cromosomi omologhi è nota come teoria cromosomica dell'ereditarietà.
La teoria cromosomica dell'ereditarietà venne confermata pochi anni dopo dal lavoro sperimentale dello statunitense T.H.Morgan e dai suoi collaboratori
Tra il 1910 ed il 1920 Thomas H.Morgan (1866 - 1945) condusse un'importante serie di esperimenti sul moscerino della frutta (Drosophila melanogaster). L'insetto si riproduce rapidamente (circa 15 giorni), genera molti discendenti, si alleva facilmente e possiede solo 4 coppie di cromosomi. I ricercatori del gruppo di Morgan sottoposero a diversi agenti mutageni i moscerini al fine di indurre la comparsa di nuovi caratteri ed incrociarono i mutanti con le varietà selvatiche, verificando le leggi dell'ereditarietà mendeliana.
A differenza del pisum sativum usato da Mendel, la Drosophila non è autofecondante e quindi per ottenere linee pure (individui omozigoti per un determinato carattere) è necessario selezionarli. In altre parole è necessario incrociare tra loro individui che presentano il carattere da selezionare, scartando ad ogni generazione gli individui che manifestano caratteri differenti fino a quando si ottengono, per più generazioni, solo individui che presentano il carattere desiderato.
I risultati degli incroci dimostrarono che non sempre gli alleli di due geni differenti segregano in modo indipendente, come aveva evidenziato Mendel. Gli alleli segregano in modo indipendente solo se i geni sono posti su coppie differenti di cromosomi omologhi. Se i due geni si trovano invece sulla stessa coppia di omologhi i loro alleli, in prima approssimazione, non possono segregare in modo indipendente. I geni che tendono a rimanere insieme, perché posti sulla stessa coppia di omologhi, si dicono appartenenti allo stesso gruppo di associazione. Il numero dei gruppi di associazione corrisponde al numero delle coppie di cromosomi omologhi.
Morgan e il suo gruppo scoprirono tuttavia che tale associazione è raramente assoluta. Le combinazioni di caratteri presenti nei genitori possono, infatti, rimescolarsi nella discendenza.
In uno dei suoi esperimenti Morgan incrociò una linea pura selvatica a Corpo grigio ed ali normali con una linea pura mutante a corpo nero ed ali atrofiche (o vestigiali). In F1 ottenne tutti ibridi a corpo grigio ed ali normali. Il colore grigio (allele G) è dominante sul colore nero (allele g) , le ali normali (allele N) sono dominanti sulle ali atrofiche (allele n).
Successivamente Morgan reincrociò i diibridi F1 con individui recessivi a corpo nero ed ali atrofiche. Ricordiamo che in questo caso i due caratteri sono associati se si ottiene una progenie con solo i fenotipi parentali (D/D e R/R) in egual proporzione, mentre i due caratteri sono indipendenti se oltre ai fenotipi parentali si ottengono in egual proporzione anche i fenotipi ricombinanti (D/R e R/D).
In F2 Morgan ottenne invece una situazione intermedia rispetto a quelle attese. Ottenne infatti tutte le combinazioni fenotipiche (D/D, D/R, R/D, R/R), ma non nella proporzione 1:1:1:1 tipica dei caratteri indipendenti. Ottenne infatti l'80% circa di individui con fenotipo parentale (D/D e R/R) ed il 20% circa di individui con fenotipo ricombinante (D/R e R/D).
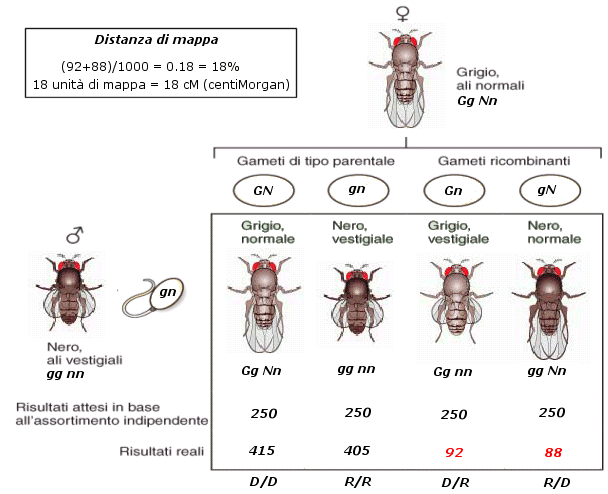
Morgan ipotizzò correttamente che i due caratteri fossero associati, ma che durante la meiosi avvenisse uno scambio fisico di materiale genetico tra le coppie di cromosomi omologhi, chiamato crossing-over (quando avviene, il crossing-over può essere osservato al microscopio, perché i cromosomi omologhi appaiati sono congiunti e assumono una struttura a X con formazione di chiasmi).

Morgan notò infine che prendendo in considerazione diverse coppie di caratteri associati, la percentuale di individui ricombinanti è diversa per ogni coppia di caratteri, ma caratteristica e costante per ciascuna coppia. Egli giustificò il fenomeno ipotizzando che il crossing-over sia un evento casuale che può avvenire con la stessa probabilità in un punto qualsiasi, lungo tutta la lunghezza del cromosoma. Di conseguenza la frequenza della ricombinazione tra due geni dipende dalla loro distanza sul cromosoma. Se i geni sono relativamente lontani vi è una maggior probabilità che il crossing-over interessi il tratto di cromosoma che li separa ed i gameti ricombinanti saranno di conseguenza frequenti, mentre se i geni sono più vicini è meno probabile che il crossing-over interessi il breve tratto di cromosoma che li separa ed i gameti ricombinanti saranno rari.
Dalla frequenza di ricombinazione tra due o più geni
diversi, misurata in esperimenti di incrocio opportunamente progettati, è
possibile dedurre quindi la distanza che intercorre tra loro su ciascun cromosoma
e costruire così delle mappe
cromosomiche o mappe genetiche. Il
posto di un carattere nella mappa genetica si chiama locus (plur. loci).
La distanza tra due geni associati si misura in Unità di mappa o centimorgan (cM).
Per definizione due geni associati distano 1 cM quando si genera 1 gamete ricombinante ogni 100 gameti (1 ogni 25 meiosi). Normalmente si usa far coincidere la distanza in centimorgan con la percentuale di ricombinazione (1 cM = 1% di ricombinazione).
La distanza di mappa è quindi uguale alla frequenza di ricombinazione scritta come percentuale.
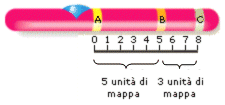
Le percentuali di ricombinazione possono essere utilizzate per individuare le posizioni relative dei geni all'interno dei cromosomi. Così se il gene A e B presentano una frequenza di ricombinazione del 5% (5 unità di mappa), il gene B e C del 3% (3 unità di mappa) ed il gene A e C dell'8% (8 unità di mappa), è evidente che il gene B si trova tra A e C.
La misura sperimentale della frequenza di ricombinazione è più semplice se si reincrociano, come abbiamo appena visto, i diibridi F1 con gli omozigoti recessivi. In questo modo, poiché l'omozigote recessivo non ha effetti sul processo di ricombinazione presentando tutti alleli recessivi, le frequenze dei genotipi ricombinanti che si ottengono dall'incrocio dipendono solo dal diibrido e sono esattamente uguali alle frequenze con cui ricombinano i gameti del diibrido, frequenze che vogliamo appunto misurare.
La misura sperimentale della frequenza di ricombinazione incrociando due diibridi è comunque possibile anche se risulta più complessa.
Se chiamiamo p la probabilità che il diibrido formi gameti parentali (AB e ab) e q la probabilità che il diibrido formi gameti ricombinanti (Ab e aB), allora la probabilità di ricombinazione dei gameti che vogliamo calcolare sarà pari a 2q.
Tenendo ora presente che incrociando i diibridi il quadrato di Punnett ci fornisce i seguenti risultati

Ricordando inoltre che gli omozigoti recessivi (aa bb) sono gli unici individui che presentano un fenotipo differente da tutti gli altri e sono quindi facilmente individuabili, possiamo misurarne la frequenza p2. Estraendone poi la radice quadrata possiamo calcolare la frequenza p con cui si formano i gameti ab e AB. Ma poiché la somma di tutte le frequenze di formazione dei gameti deve dare l'unità (p + p + q + q = 2p + 2q = 1), possiamo calcolare la frequenza di ricombinazione 2q sottraendo 2p all'unità
2q = 1 - 2p
Ad esempio,
incrociando i diibridi F1 di Drosofila con corpo grigio ed ali
normali, avremmo ottenuto in F2 un 16.8 % (frequenza = 0,168) di
individui, recessivi per entrambi i caratteri, a corpo nero ed ali atrofiche.
Si ha quindi p2 = 0,168 e dunque p = ![]() = 0.41.
= 0.41.
La frequenza di ricombinazione è allora 2q = 1 - 2p = 1 - 2(0.41) = 1 - 0.82 = 0.18 pari al 18%
 |
|
| Appunti Geografia |  |
| Tesine Ingegneria tecnico |  |
| Lezioni Biologia |  |